本ページではFreeSurfer 7.0でリリースされた海馬・扁桃体のセグメンテーションを行うモジュールについて解説します。
1. Recon-allされたT1強調画像のみを使用
2. 複数の画像データを用いる(ex. T1WIに加えてT2WIを追加する)
3. T1強調画像の縦断的解析を行う
本ページでは1について記載します。予めT1WIを用いたRecon-all後のデータが必要になりますので、FreeSurferのインストール及びRecon-allまでの手順に関してはこちらをご確認ください。
また、MacOSのupdateでデフォルトシェルがzshになっている方は、bashに戻しておきましょう。下記コマンドを入力の上でターミナルを再度立ち上げてください。
chsh -s /bin/bash
インストール
海馬モジュールはFreeSurfer7.0に搭載されているため、追加のインストールは必要ありません。しかし、モジュール使用のためにMatlabR2014bランタイムが必要になります。下記コマンドをターミナルに入力してください。
fs_install_mcr R2014b
fs_install_mcrが使用できない場合は下記コマンドも試してみてください。
cd $ FREESURFER_HOME / bin && curl https://raw.githubusercontent.com/freesurfer/freesurfer/dev/scripts/fs_install_mcr -o fs_install_mcr && chmod + x fs_install_mcr
また、権限の問題でインストールが上手くいかない際は、インストール先であるusr/local/freesurfer/7.1.1 ディレクトリを対象にchmodコマンドを使って権限を付与しましょう。
セグメンテーションの実行
次に海馬・扁桃体セグメンテーションを行います。Recon-allを実行済みのサブジェクトを用意してください。今回はtest001というサブジェクトを使用します。
cd $SUBJECTS_DIR
segmentHA_T1.sh test001
少し時間はかかりますが、下記のように表示されれば成功です。
freesurfer > subjects > test001 > mri 内に関連ファイルが出力されていることを確認しましょう。
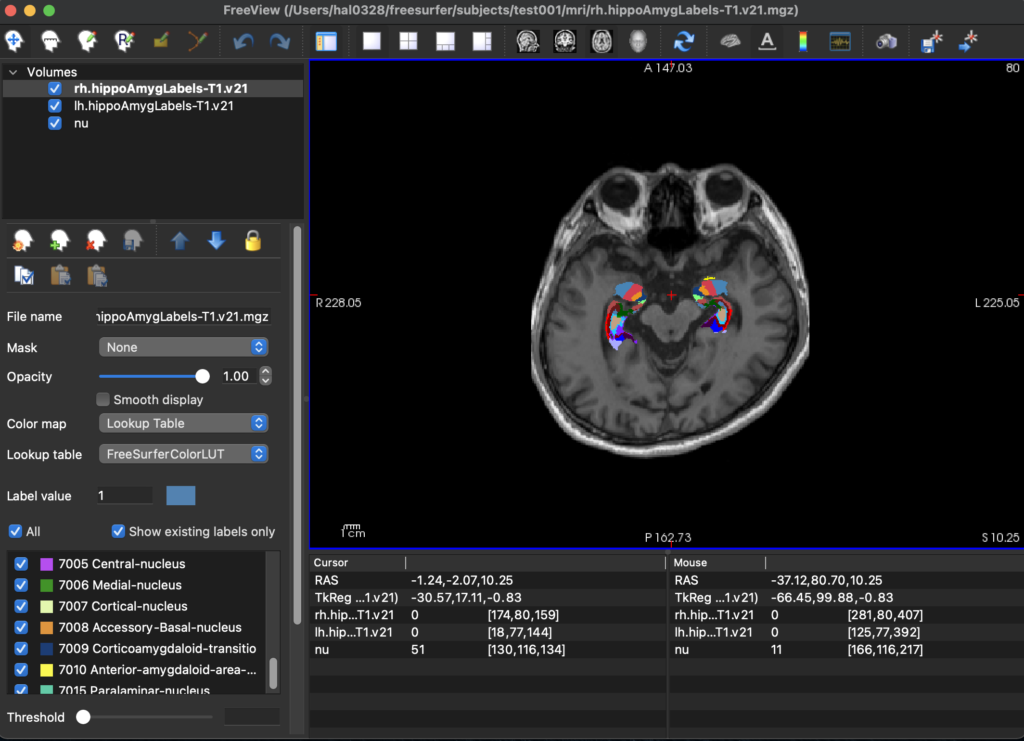
セグメンテーションの確認
[lh/rh].hippoAmygLabels-T1.v21.mgzに0.333mmボクセルのセグメンテーション情報が保存されています。また、[lh/rh].hippoAmygLabels-T1.v21.[hierarchy].mgz ([hierarche]=HBT, FS60, CA)に上述した異なるセグメンテーションが保存されています。
freeviewを用いてセグメンテーションされた画像データを確認できます。
cd $SUBJECTS_DIR
freeview -v \
test001/mri/nu.mgz \
test001/mri/lh.hippoAmygLabels-T1.v21.mgz:colormap=lut \
test001/mri/rh.hippoAmygLabels-T1.v21.mgz:colormap=lut
HBTセグメンテーション
cd $SUBJECTS_DIR
freeview -v \
test001/mri/nu.mgz \
test001/mri/lh.hippoAmygLabels-T1.v21.HBT.mgz:colormap=lut \
test001/mri/rh.hippoAmygLabels-T1.v21.HBT.mgz:colormap=lut
FS60セグメンテーション
cd $SUBJECTS_DIR
freeview -v \
test001/mri/nu.mgz \
test001/mri/lh.hippoAmygLabels-T1.v21.FS60.mgz:colormap=lut \
test001/mri/rh.hippoAmygLabels-T1.v21.FS60.mgz:colormap=lut
CAセグメンテーション
cd $SUBJECTS_DIR
freeview -v \
test001/mri/nu.mgz \
test001/mri/lh.hippoAmygLabels-T1.v21.CA.mgz:colormap=lut \
test001/mri/rh.hippoAmygLabels-T1.v21.CA.mgz:colormap=lut
[lh/rh].hippoSfVolumes-T1.v21.txtには海馬の体積量が、[lh/rh].amygNucVolumes-T1.v21.txtには扁桃体の体積量が記録されています。
追記(2022.7.29): Freesurfer 7.1.1のHippocampalSubfieldsAndNucleiOfAmygdalaにおいて、Label 231-246と解剖学的部位との対応がFsTutorialAnatomicalROIFreeSurferColorLUTに記載されていないようです。4つのラベルの出力内容の違いについては公式サイトをご参照ください。
203 parasubiculum
204 presubiculum
205 subiculum
206 CA1
208 CA3
209 CA4
210 GC-DG
211 HATA
212 fimbria
214 molecular_layer_HP
215 hippocampal_fissure
226 HP_tail
231 HP-body
232 HP-head
233 presubiculum-head
234 presubiculum-body
235 subiculum-head
236 subiculum-body
237 CA1-head
238 CA1-body
239 CA3-head
240 CA3-body
241 CA4-head
242 CA4-body
243 GC-ML-DG-head
244 GC-ML-DG-body
245 molecular_layer_HP-head
246 molecular_layer_HP-body
7001 Lateral-nucleus
7003 Basal-nucleus
7005 Central-nucleus
7006 Medial-nucleus
7007 Cortical-nucleus
7008 Accessory-Basal-nucleus
7009 Corticoamygdaloid-transitio
7010 Anterior-amygdaloid-area-AAA
7015 Paralaminar-nucleus










コメント