今回は脳MRIから3Dモデリングを作成する方法について解説します。
研究発表のスライドを作成する際に、神経回路や特定の領域を示すための図を載せたい、スケルトンの3Dモデリングを自分で扱うことができたら、と考えて久しぶりに執筆しています。
3Dモデリングの処理にはBlenderを利用します。Blenderの操作に慣れを要するかもしれませんが、本記事ではBlenderへの出力までを扱いますので、ソフトの操作については別サイトを参照ください。
MRIからNifti形式に変換
撮像されたMRIはDICOM形式と思いますので、dcm2niiなどを使用してNifti形式に変換してください。
必要に応じて下記記事も参照ください。

Niftiファイルをrecon-allして脳画像を作成
Blenderにインポートするために、NiftiをSTLやOBJ形式に変更する必要があります。ただし撮像したMRIは脳以外の組織(骨・軟部組織)も含まれており、脳以外の領域を除外する必要があります(≒Skull Stripping)
MRIcroGLや3D slicerを用いて自身でSkull Strippingする方法もありますが、用手的なSegmentationは煩雑なため、FreeSurferでRecon-allしたデータを用いる方法を採用しています。
FreeSureferの使用方法は下記ページを参考にしてください。

以下のコードを使用して、Recon-allで出力されたaseg.mgzから、両側大脳皮質、小脳、脳幹を指定し、結合したファイルを作成します。今回は”test001″というSubjectを対象として処理しています。各々のSubject nameに変更してください。
mri_binarize --i $SUBJECTS_DIR/test001/mri/aseg.mgz \
--match 2 3 7 8 16 41 42 46 47 \
--o $SUBJECTS_DIR/test001/mri/aseg_whole_brain.mgz作成した脳画像から mri_mcコマンドを使用して表面メッシュ画像を作成します。mri_mcは、指定されたラベル値に基づいてメッシュを生成します。したがって、aseg_whole_brain.mgzに含まれる領域ラベルの番号を指定する必要があり、全脳を対象とするために、”1″を指定します。
mri_tessellate $SUBJECTS_DIR/test001/mri/aseg_whole_brain.mgz 1 \
$SUBJECTS_DIR/test001/surf/whole_brainここで生成したメッシュ画像はVoxel単位のセグメンテーションを利用しているため、表面がつぶつぶと角ばったものになってしまいます。表面を平滑化するために下記のコードを使用します。平滑化の回数は5回を指定しています。
mris_smooth -n 5 $SUBJECTS_DIR/test001/surf/whole_brain \
$SUBJECTS_DIR/test001/surf/whole_brain_smooth最後に出力されたwhole_brain_smoothというメッシュ画像を、Blenderに読み込ませるためにSTLファイルへ変更します。
mris_convert $SUBJECTS_DIR/test001/surf/whole_brain_smooth \
$SUBJECTS_DIR/test001/surf/whole_brain_smooth.stlBlenderでSTLファイルを読み込む
まだの方は Blender をインストールしましょう。初期設定で問題ありません。
メインウィンドウが開いたら、STLファイルをdrag & dropで読み込むことができます。
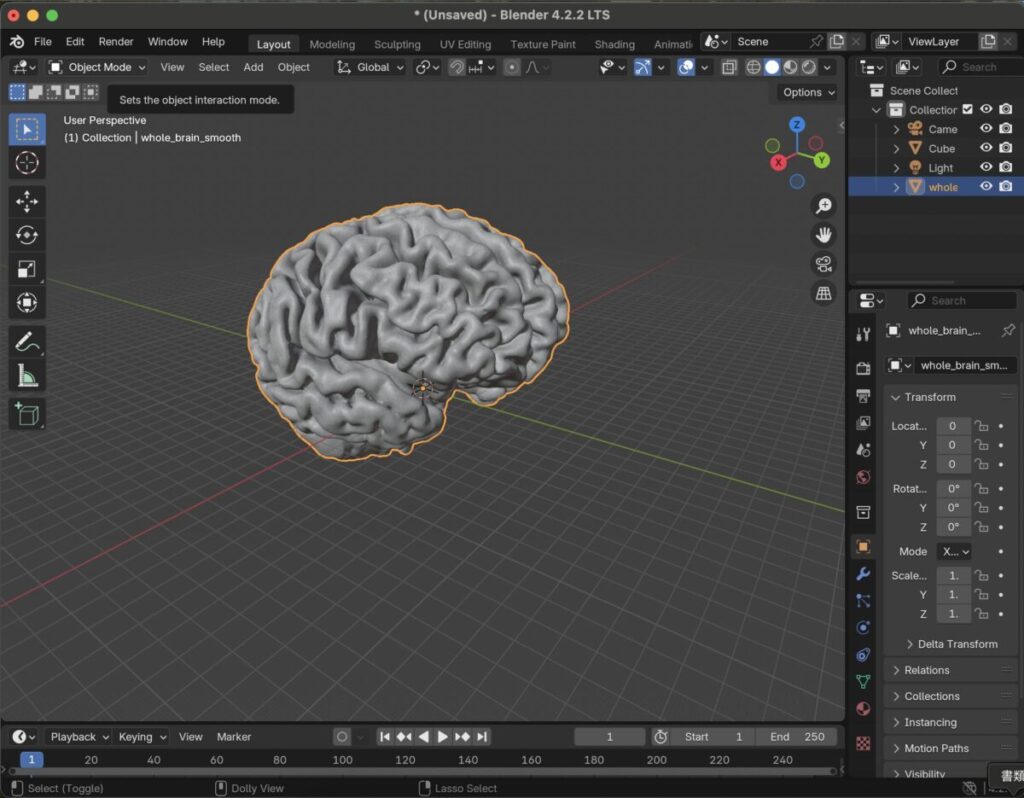
平滑化前の画像
平滑化後の画像
これで、脳MRIからBlenderで操作できる3Dオブジェクトに変換することができます。
MRIからSkullなど周辺組織を除外するハードルが高いため、FreeSurferを用いる方法が最もリーズナブルだと感じました。










コメント