前ページでは下記リストの1-2まで完了しました。
本ページでは 3 の出力データ生成について解説します。
今回はFSDGファイルを参照し、[ mris_preproc ] 及び [ mri_surf2surf ]コマンドを用いて
サブジェクトファイルを1つのファイルに連結すること、
[ mri_glmfit ]コマンドを用いて線形モデルにフィッティングを行い解析すること、の2つを行います。
前ページ:【FSGD file】FreeSurferを用いたSurface group analysis #1

今回の流れです。
- FSGDファイル作成
- コントラストファイルの作成
- mris_preproc によるリサンプリング
- mri_surf2surf によるデータの平滑化
- mris_glmfitを使用した線形モデルのフィッティング
- Freeviewで出力の確認
下記サイトを参考にして記載しています。
mris_preproc によるグループファイル作成 (リサンプリング)
まず下記のコマンドで [ mris_preproc ]を行い、サブジェクトを連結して一つのファイルを作成します。
この状態は平滑化される前であり、使用するサブジェクトは fsgd fileによって指定されます。
mris_preproc –fsgd TestStudy.fsgd \
–target fsaverage \
–hemi lh \
–meas thickness \
–out lh.TestStudy.thickness.00.mgh
–fsgd XX.fsgd :自身のfsgd fileのファイル名を入力してください。
–hemi XX:右半球 or 左半球は rh or lh のどちらかを入力してください。
–meas XX:計測する内容を入力してください。
–out XX.mgh:自身の使用するStudy名や計測内容を指定してください。
上記コマンドでは左半球の皮質厚を fsaverage にリサンプリングする形で出力しています。
mri_surf2surf によるデータの平滑化
次に[ mri_surf2surf ]を用いてリサンプリングされたデータを平滑化します。下記のコマンドを用いてください。
mri_surf2surf –hemi lh \
–s fsaverage \
–sval lh.TestStudy.thickness.00.mgh \
–fwhm 10 \
–cortex \
–tval lh.TestStudy.thickness.10.mgh
–sval XX:リサンプリングに使用したファイル名を指定してください。
–fwhm XX:何mm FWHM : Full Width at Half Maximum で平滑化を行うか設定してください。
–cortex:皮質のみを設定しています。他のラベルの設定も可能です。
–tval:出力するファイル名です。入力ファイル名から 00→ XX (fwhm)に変更してください。
lh.TestStudy.thickness.10.mgh が出力されれば平滑化は完了です。
mris_glmfit を使用した線形モデルのフィッティング
General linear model (GLM) フィッティングは [ mris_glmfit ]コマンドを使用して行います。
mri_glmfit \
–y lh.TestStudy.thickness.10.mgh \
–fsgd TestStudy.fsgd dods \
–C Contrast1GroupDiff.mtx \
–surf fsaverage lh \
–cortex \
–glmdir lh.TestStudy.glmdir
–y XX:入力ファイル名です。
–fsgd XX dods:デフォルトでDODSが設定されています。詳細はこちらで説明します。
–C XX:コントラストファイルの設定です。複数のファイルが指定できます。
–glmdir XX:出力ディレクトリの名称設定です。
エラーがなければ2,3分で解析が終わります。
lh.TestStudy.glmdir ディレクトリが作成されたら下記コマンドで内容を確認しましょう。
ls lh.TestStudy.glmdir
次に下記コマンドで重要度マップをFreeviewを用いて表示しましょう。
lh.TestStudy.glmdir における Contrast1GroupDiff を参照し、
グループインターセプト間の有意差について検証しています。
具体的には「年齢と性別の影響を除外し、グループインターセプト間に有意差はない」という帰無仮説に対して、
t検定を行っています。
freeview -f $SUBJECTS_DIR/fsaverage/surf/lh.inflated:annot=aparc.annot:annot_outline=1:overlay=lh.TestStudy.glmdir/Contrast1GroupDiff/sig.mgh:overlay_threshold=4,5 -viewport 3d -layout 1
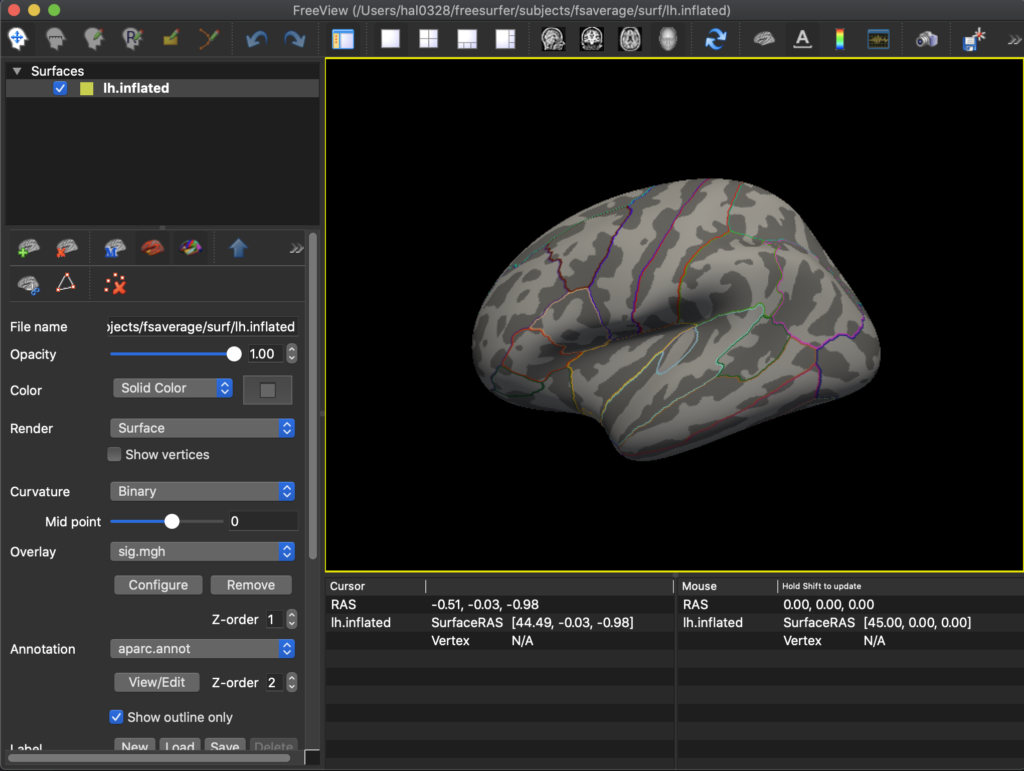
左下、Overlay内にあるConfigureをクリックすると、比較した領域を描画する閾値の設定ができます。
上記のコマンドで初期の閾値の設定は 4-5となっていますが、だいぶ厳しい設定なので適宜変更してください。
Configure内の設定はあくまでGUI上の描画のためのものであり、多重比較を行うことでクラスターを決定できます。
多重比較のためのクラスター修正
FreeSurferではクラスターの修正を行うためにシミュレーションを実行します。
シミュレーションではクラスターサイズが最大になる測定値を取得した上で多重比較を行います。
下記コマンドをターミナルに入力してください。
mri_glmfit-sim \
–glmdir lh.TestStudy.glmdir \
–cache 4 neg \
–cwp 0.05\
–2spaces
–cwp 0.05:p値が0.05未満のクラスターを保持するよう設定しています。
コントラストファイル内にファイルが作成されていることが確認できます。
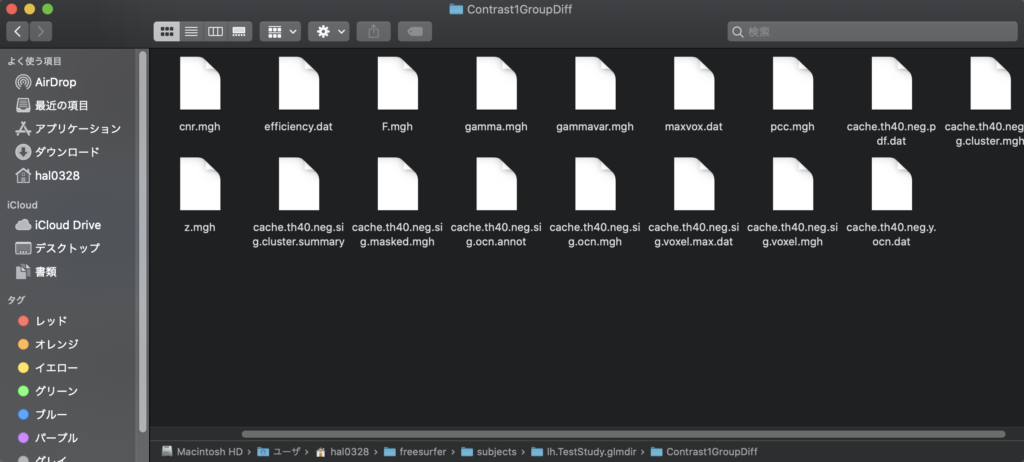
各ファイルの内容は以下の通りです。
- cache.th40.neg.sig.cluster.mgh :多重比較後のクラスターごとのマップ
- cache.th40.neg.sig.cluster.summary :クラスターの要約
- cache.th40.neg.sig.masked.mgh :多重比較後も存続する未修正のsig値
- cache.th40.neg.sig.ocn.annot :出力クラスター番号(クラスターの注釈)
- cache.th40.neg.sig.ocn.mgh :各クラスターの場所を示すセグメンテーション
- cache.th40.neg.sig.voxel.mgh :多重比較のために修正されたボクセルマップ
- cache.th40.neg.y.ocn.dat :各クラスター内の各サブジェクトの平均値
下記のコマンドで 3.クラスターの要約を確認してみましょう。
less lh.TestStudy.glmdir/Contrast1GroupDiff/cache.th40.neg.sig.cluster.summary
以下のコマンドで、各クラスターをFreeview上で確認できます。
freeview -f $SUBJECTS_DIR/fsaverage/surf/lh.inflated:overlay=lh.TestStudy.glmdir/Contrast1GroupDiff/cache.th40.neg.sig.cluster.mgh:overlay_threshold=2,5:annot=lh.TestStudy.glmdir/Contrast1GroupDiff/cache.th40.neg.sig.ocn.annot -viewport 3d
脳表に色分けされた領域が各クラスターです。
クラスターをクリックするとクラスター番号と数値が表示されます。数値は log10-となっています。
マイナスの値は負の相関を示します。
クラスターが表示されない場合は、閾値を(ex cache=3 or 2 or 1.3…)下げてみてください。
surf/lh.inflatedの部分をlh.pialに変更し、脳表画像上にクラスターを表示させることができます。
論文に使用する際はこの形式で用いることが多いようです。
freeview -f $SUBJECTS_DIR/fsaverage/surf/lh.inflated:overlay=lh.TestStudy.glmdir/Contrast1GroupDiff/cache.th40.neg.sig.cluster.mgh:overlay_threshold=2,5:annot=lh.TestStudy.glmdir/Contrast1GroupDiff/cache.th40.neg.sig.ocn.annot -viewport 3d
これでFreeSurferを用いたSurface group analysisは終了です。お疲れ様でした。










コメント