前ページ:【FreeView①】FreeSurferインストール後に行うべきこと #2
前回は Recon-all 後の Subject が正確に前処理できているか、freeviewを用いて確認しました。
今回は皮質のParcellationについて、別のコマンドでfreeviewから確認します。
freeview -f test001/surf/lh.pial:annot=aparc.annot:name=pial_aparc:visible=0 \ test001/surf/lh.pial:annot=aparc.a2009s.annot:name=pial_aparc_des:visible=0 \ test001/surf/lh.inflated:overlay=lh.thickness:overlay_threshold=0.1,3::name=inflated_thickness:visible=0 \ test001/surf/lh.inflated:visible=0 \ test001/surf/lh.white:visible=0 \ test001/surf/lh.pial \ --viewport 3d
例に漏れず、[ test001 ] は自身のsubject名に置換してください。
コマンドを入力すると以下のようにfreeview が立ち上がります。
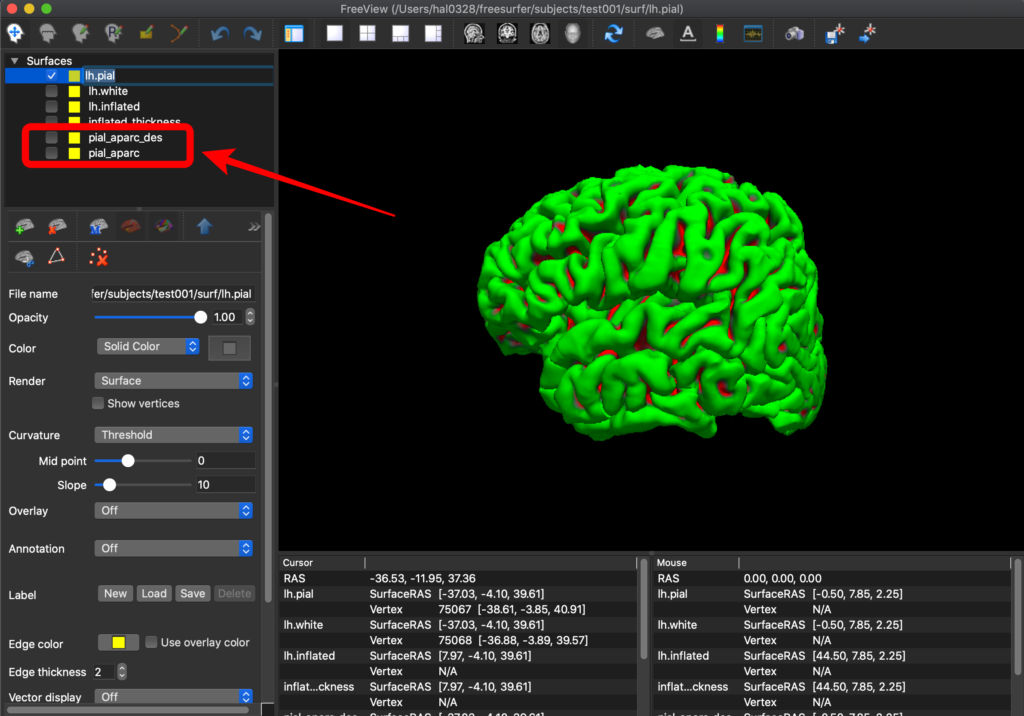
皮質を再構成した3D viewの lh : left hemisphere が表示されています。[ lh.pial ] のチェックを外し、
図中・赤矢印の [ pial_aparc_des ] または [ pial_aparc ] にチェックを入れて、
皮質のParcellationが行われていることを確認してください。
[ pial_aparc_des ]は Destrieux アトラスで、
[ pial_aparc ]は Desikan-Killiany アトラスで作成された皮質 Parcellation 画像です。
下記のような画像を確認できます。
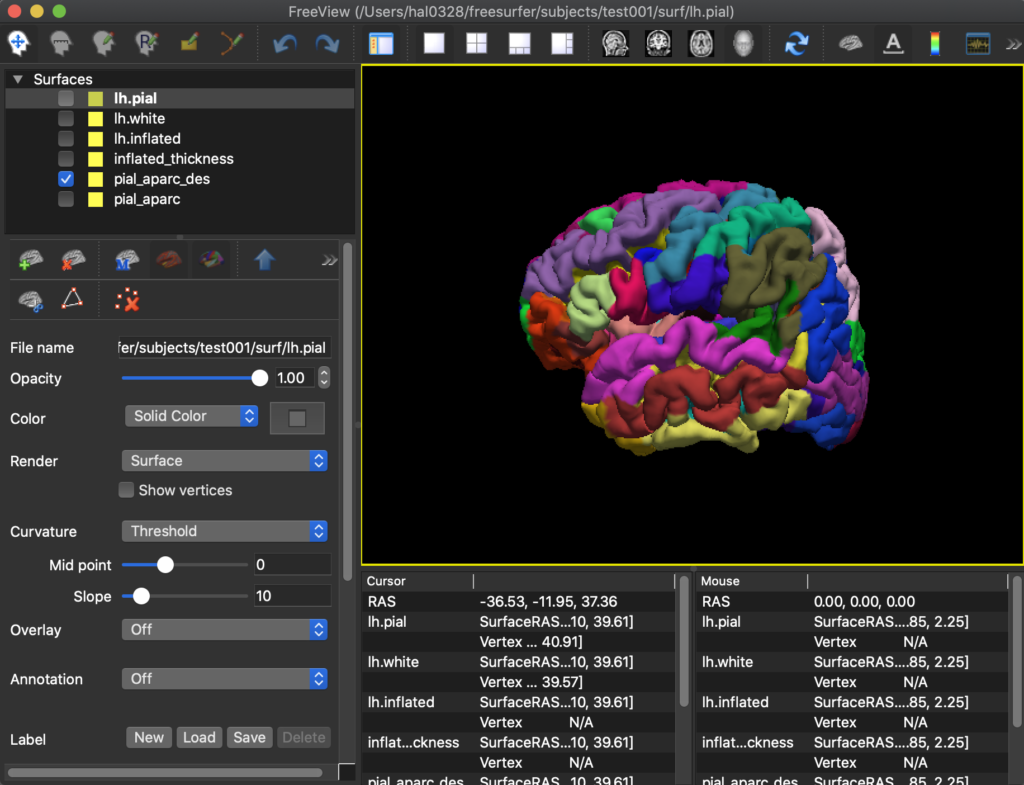
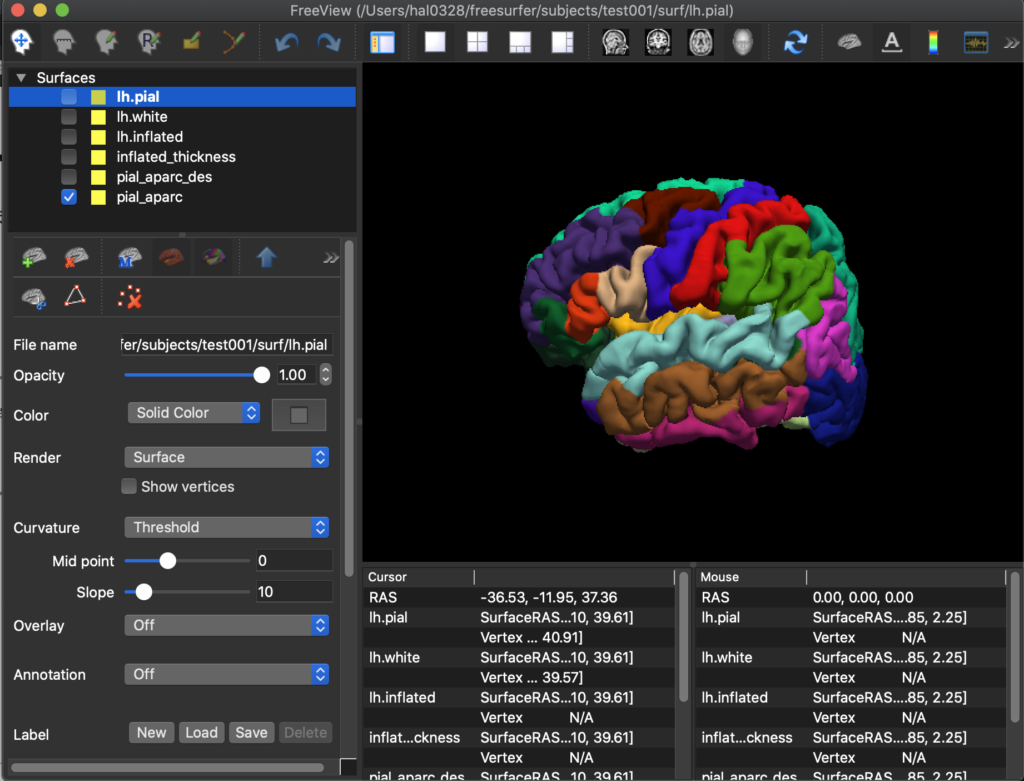
皮質のTopological defects を修正する方法については別ページで説明します。
本家サイトのこちらも確認してください。










コメント
コメント一覧 (3件)
HAL先生
本サイトにてFreeSurferの利用方法について、学ばせていただいております。
SATと申します。
FreeSurferインストール後に行うことを順当に実施できたのですが、
本ページの冒頭に出てくる皮質のPercellationを確認するためのコマンドを入力したところ、Failed to load Surface となってしまい、Window左上のSurfacesには、pail_aparcのチェックボックスのみ表示されます。Surfaceの読み込みを正しく行うために、アドバイス頂けましたら幸いです。
返信が遅くなり、大変申し訳ございません。
webサイトの設計自体を変更したことで、表示されているスクリプトのcopy&pasteがうまく行かないようです。
元サイトのスクリプトや、本ページに追記したスクリプトでは表示できると思いますので、ご確認頂けますと幸いです。
HAL先生、ご返信いただきありがとうございます。
こちら返信遅くなり、申し訳ございません。
スクリプトの追記をしていただき、ありがとうございました。
無事に表記されました。元サイトも参考に、試して参りたいと思います。
ありがとうございます。