本ページではFreeSurferにおいてセグメンテーションされた体積やパーセレーションされた皮質厚・皮質表面積などを
Tableとして出力する方法を説明します。
Subjectの生成方法については前ページ
【Recon-all】FreeSurferインストール後に行うべきこと #1 を参照してください。

統計ファイル作成のTutorialは下記サイトを参考にしています。
Subject 内の stats フォルダには、ラベル画像から作られた複数の統計ファイルが入っています。
Segmentation volume to table
セグメンテーションされた各領域の体積(mm3)は下記のコマンドで Tableにできます。
cd $SUBJECTS_DIR
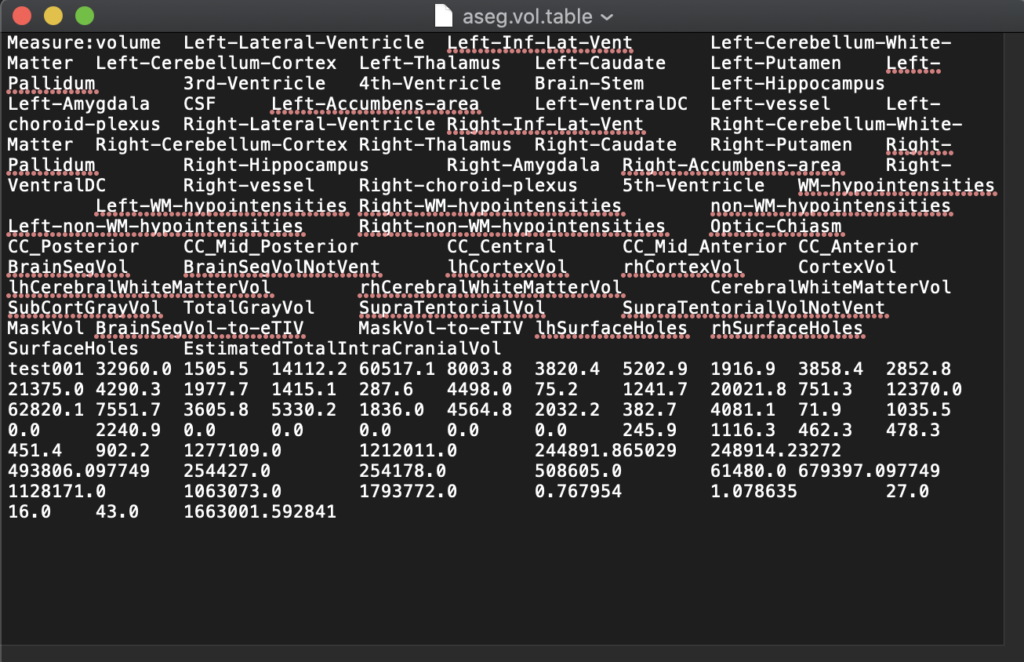
asegstats2table –subjects test001 –tablefile aseg.vol.table
asegstats2tableコマンドは、解析したSubjectのセグメンテーション体積からtableを作成します。
デフォルトではHomeフォルダ内に aseg.vol.table が作成されています。
CSV形式で保存されているため、開けない場合は下記コマンドを入力しテキストソフトで開きましょう。
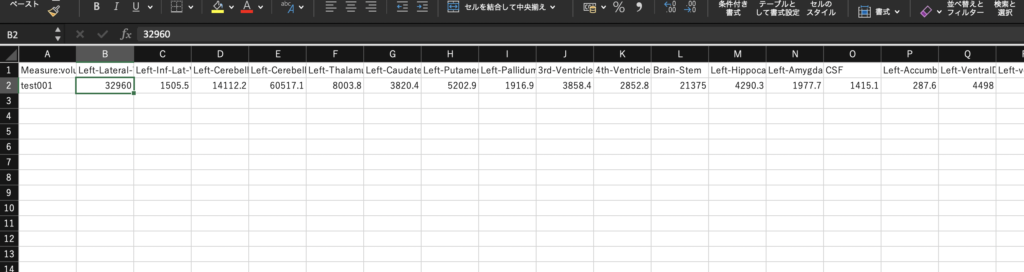
Excel にコピー&ペーストすると Table にできます。
open -e aseg.vol.table
[ –subjects XXX YYY ZZZ ] に該当する部分には対象となるサブジェクト名を入力してください。
複数のサブジェクトを半角スペースで区切って入力することで、一つのTable に纏めることができます。
[–segno XX YY ZZ ] を加えることで、特定の領域に限定して Tableにすることができます。
領域は入力する数字によって決まるため、FreeviewもしくはLabel numberから該当する数字を検索してください。
上記を踏まえて、解析に必要な形でコマンドを設定してください。下記はその一例です。
cd $SUBJECTS_DIR
asegstats2table –subjects test001 test002 test003 \
–segno 11 17 18 \
–tablefile aseg.vol.table
open -e aseg.vol.table
各 Number とLabelの対応は下記のサイトで確認できます。
Cortical parcellation to table
パーセレーションされたデータを Table にする場合は以下のコマンドを使用します。
設定内容がSegmentation volume に比べて多いため、内容を分割して説明します。
aparcstats2table
–subjects 【①subject名】 \
–hemi 【②右半球or左半球】 –meas 【③計測内容】 –parv 【④アトラス設定】 \
–tablefile 【⑤ファイル名】
- Subject名はSegmentation volumeの際と同様で、自身のサブジェクト名を入力してください。
複数のサブジェクトを同時に解析することも可能です。 - 大脳半球の左右どちらかを指定します。[ lh ] or [ rh ]を入力してください。
- 計測内容はデフォルトで表面積に設定されています。
他に Volume:体積、thickness:皮質厚、thicknessstd:皮質厚の標準偏差、meancurv:平均曲線、
gauscurv:ガウス曲率、foldind:折りたたみインデックス、 curvind:曲率インデックス が入力できます。 - アトラス設定です。デフォルトでは aparc : Desikan-Killiany Atlas が設定されています。
aparc.a2009s と入力した場合 Destrieux Atlas に変更できます。 - ファイル名です。大脳半球 + Atlas + 計測内容とよく設定されます。
これらを用いて必要なコマンドを設定し、サブジェクトから Tableファイルを作成してください。
下記はその一例で、lh.aparc.a2009.thickness.tableというTable名に設定し、
内容は 左半球+Destrieux Atlas+皮質厚が出力されるようにします。
cd $SUBJECTS_DIR \
aparcstats2table –subjects test001 test002 test003 \
–hemi lh –meas thickness –parc aparc.a2009s \
–tablefile lh.aparc.a2009s.thickness.table










コメント
コメント一覧 (8件)
【FreeSurfer】サブジェクトから統計ファイルを作成する方法の中で、
Segmentation volume to tableのところは問題なく、各領域の体積が表示されたのですが、Cortical parcellation to tableのところは、
cd $SUBJECTS_DIR \
aparcstats2table –subjects test001 test002 test003 \
–hemi lh –meas thickness –parc aparc.a2009s \
–tablefile lh.aparc.a2009s.thickness.table
のコマンドを入力しても、lh.aparc.a2009.thickness.tableというTableがどこに保存されたのかがよく分かりませんでした。
open -e lh.aparc.a2009s.thickness.table
と入力しても該当する”ファイルが存在しない”とメッセージがでます。
どうしたら良いでしょうか?
問題なく解析ができておりましたら、
***
SUBJECTS_DIR : /Users/XX/freesurfer/subjects
Parsing the .stats files
Building the table..
Writing the table to lh.aparc.a2009s.thickness.table
XX-MacBookPro:subjects XX$
(XX:ユーザ名)
***
の様な表示が出て、subjectディレクトリ内にtableファイルが作成されるはずですが、
Errorメッセージなどは出ておりませんでしょうか?
また、test001-003は仮のサブジェクト名ですが、
Recon-all後のファイル自体には問題ありませんでしょうか?
ご返信いただきありがとうございます。
ご記載いただいたような表示は出ず、
XXXs-MacBook-Pro:subjects XXX$
(XX:ユーザ名)と表示されます。Errorメッセージは出ません。
test001の部分は、自分でつけたnc1などの名前に置き換えて入力していました。
Recon-all後にこちらのページの方法を行なうと、御記載どおりの画像が表示できてはいます。
https://nshalnote.com/?p=118
承知いたしました。XXX$と戻っているのでしたら、current directryをsubjectに設定したものの
aparcstats2tableのコマンドが反映されていない様ですね。
Freesurferが同versionでしたらコマンド自体は問題ないはずですが、
私もcopy&pasteでコマンドが反応しなかったことがあります。
その際は改行か半角が適切でなかった様に記憶しておりますので
お手数をお掛けして恐縮ですが、subjectをcurrent directryに設定後、
一度手打ちで下記内容を入力してみて頂けないでしょうか。
(Subject名は適宜変更してください。)
aparcstats2table –subjects test001 test002 test003 –hemi lh –meas thickness –parc aparc.a2009s –tablefile lh.aparc.a2009s.thickness.table
その後、表示されたメッセージを参考に下記のように打ち込みましたところ、うまく表示されました。
aparcstats2table –hemi lh –subjects nc001 –parc aparc.a2009s –meas thickness -t lh.a2009s.thickness.txt
どうもありがとうございました。
画像解析の勉強にあたり、いつもページを拝読しております。
よろしければ、ひとつ教えていただきたいことがございます。
Freesurferでは各脳葉の皮質容積、例えば前頭葉皮質の総容積というかたちでは出力されませんね。
前頭葉皮質の総容積を求めたければ、それぞれの数値を足していく必要があるのでしょうか。
total frontal lobe volumeなどのかたちで出力できる方法をご存じであれば、ご教示いただけないでしょうか。
初歩的な質問で恐縮ですが、どうぞよろしくお願いいたします。
ご連絡ありがとうございます。
脳皮質の容積、皮質厚の計算はそれぞれのAtlasにおけるROIごとに出力されてしまいます。
脳葉ごとの総容積を求めるためには個々のROIごとの数値を足す以外に、
各ROIのラベルを抽出し、脳葉ごとのラベルを作成する方法がwikiに紹介されておりました。
下記にURLを添付致します。
https://surfer.nmr.mgh.harvard.edu/fswiki/CorticalParcellation
参考になりましたら幸いです。お困りのことがございましたら再度ご連絡頂けますと幸甚に存じます。
早速ありがとうございます。
こちらの内容に沿って、解析してみます。
うまく行きましたらご報告申し上げます。